iGEM TSUKUBA LEAPS
タンパク質設計のためのWebアプリ
iGEM TSUKUBA LEAPSとは
iGEM TSUKUBA Editorは、筑波大学の一般学生団体であるiGEM TSUKUBAが作成したタンパク質設計のためのWebアプリです。 私たちが、研究している手法であるLEAPS を用いて、タンパク質設計を行うためのインターフェースを提供します。
開発背景
タンパク質工学は、創薬や育種など、様々な分野で重要な役割を果たしています。 しかし、従来のタンパク質工学は、多くのデータを必要です。 そこで、わずか数十件のデータからタンパク質設計を行うことができるLEAPSを開発しました。 しかし、このLEAPSを導入するには2つの問題点を抱えていました。
1. 導入の難しさ
LEAPSを導入するには、機械学習の知識が必要であり、多くの人にとっては敷居が高いものでした。
特に、生命科学の研究者にとっては、機械学習の知識がない場合が多く、LEAPSの導入に苦労することが予想されました。
さらに、LEAPSを運用するには、十分な計算資源が必要でした。
2. 運用の難しさ
LEAPSを運用する上で、有害なタンパク質を設計するといった不適切な使用を防ぐ必要がありました。
特に、LEAPSを開発した私たちは、倫理的な観点から、LEAPSの使用を適切に監督する必要がありました。
そこで、より多くの人にLEAPSを利用してもらうために、 iGEM TSUKUBA LEAPSでは、次の3つの機能を実装することで、これらの課題を解決することを目指しました。
主な機能
1. 専門知識が必要ないインターフェース
この機能は、ユーザーフレンドリーなインターフェースをWebアプリケーションとして提供することで、専門知識がなくてもLEAPSを導入できるようにし、「導入の難しさ」を解決します。
-
ステップ1
LEAPSを用いて有害なタンパク質を設計するといった不適切な使用を防ぐために、フォームに回答します。
-
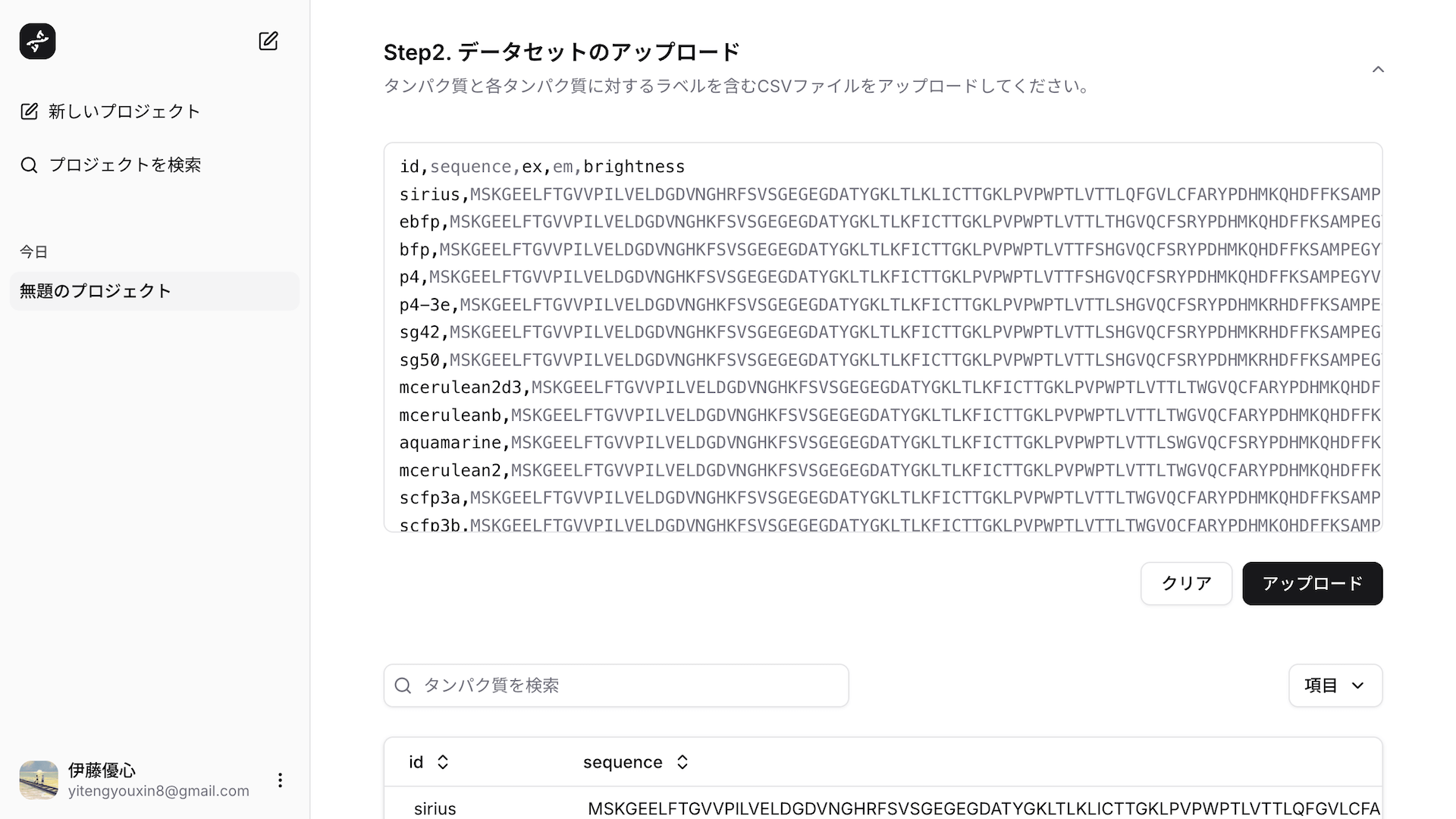
ステップ2
タンパク質と各ラベルのデータセットをアップロードします。CSV形式およびTSV形式に対応しています。
-
ステップ3
各ラベルの目的関数を設定します。最大化・最小化・範囲に対応しています。
-
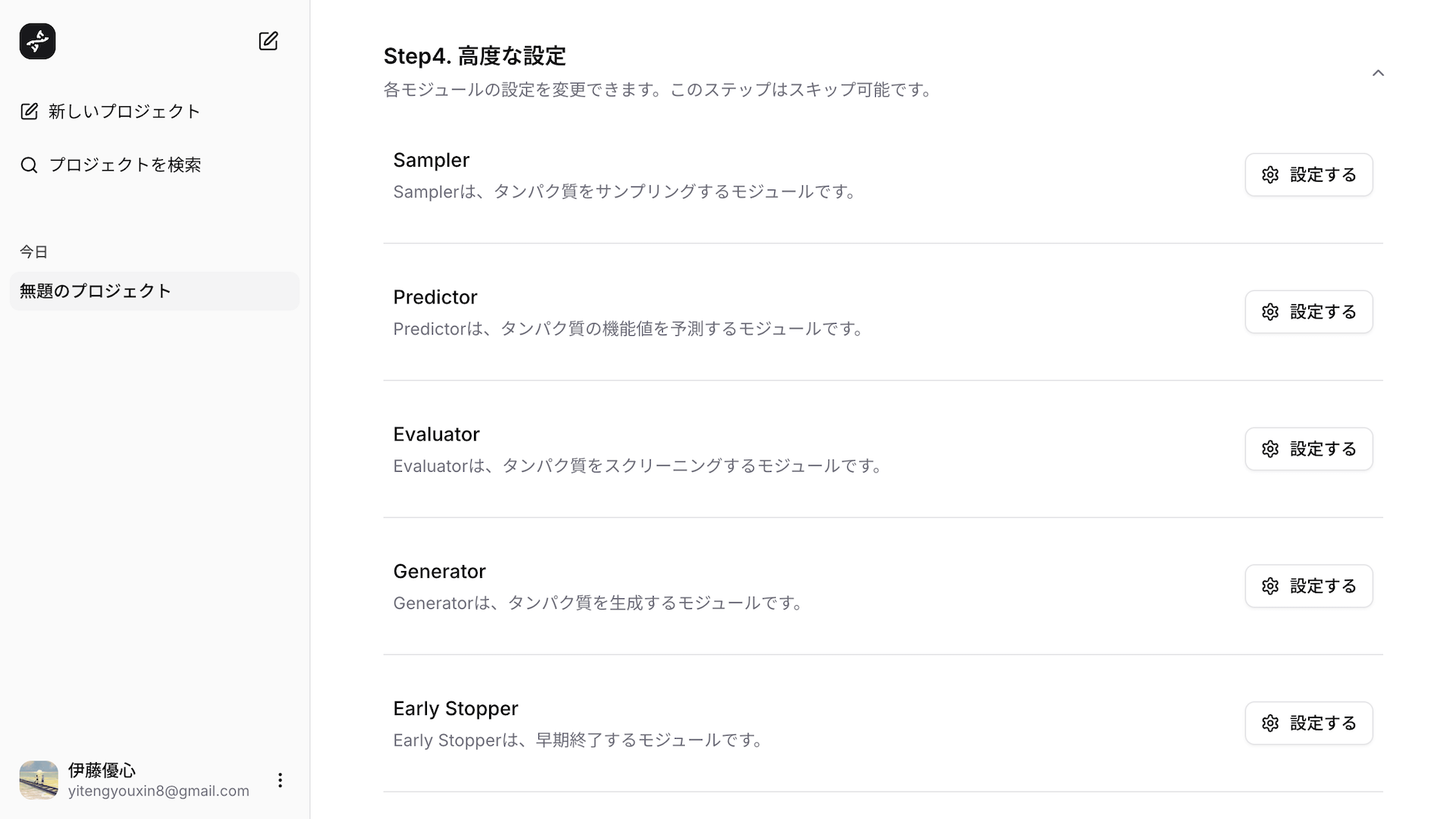
ステップ4
LEAPSが用いる各モジュールのハイパーパラメーターを調整できます。この設定は任意です。
2. 毒性タンパク質のためのデータベース
この機能は、有害なタンパク質を設計するといった不適切な使用を防ぐために、 毒性タンパク質のデータベースを用意し、設計されたタンパク質が毒性タンパク質に類似していないかを確認することで、「運用の難しさ」を解決します。
LEAPSは、以下の2つで毒性タンパク質の確認を行います。
MMSeqs2によるクラスタリング
MMSeqs2は、タンパク質の相同性に基づいてクラスタリングを行うことができるツールです。
これを用いて、毒性タンパク質と相同性が高いタンパク質を発見します。
Foldseekによるクラスタリング
Foldseekは、タンパク質の立体構造に基づいてクラスタリングを行うことができるツールです。
これを用いて、毒性タンパク質と立体構造が類似しているタンパク質を発見します。
使用した技術
Table of Contents